Region 4 Stork系统在串联质谱新生儿极长链酰基辅酶A脱氢酶缺乏症筛查中的应用
2020年10月
中华内分泌代谢杂志,第43卷第10期 第978页-第983页
张超,胡真真,杨建滨,尚世强,黄新文,杨茹莱,胡凌微,张玉,吴鼎文,赵正言
极长链酰基辅酶A脱氢酶缺乏症(very long chain acyl-CoA dehydrogenase deficiency,VLCADD)是一种较罕见的脂肪酸代谢病,由极长链酰基辅酶A脱氢酶(VLCAD)基因缺陷引起,呈常染色体隐性遗传,是婴儿期发生潜在猝死的疾病之一[1]。开展包括VLCADD在内的新生儿筛查可使患儿在尚未出现临床表征或症状轻微时及早诊治,避免机体产生不可逆的智力低下、器质性病变甚至死亡等不良后果。目前国内部分实验室采用串联质谱技术(tandem mass spectrometry,MS/MS)进行新生儿疾病筛查[2, 3],现普遍存在假阳性率偏高、召回工作量大等问题[4],在一定程度上造成医疗资源的浪费及给疑似患儿家庭增添经济负担和精神压力。
Region 4 Stork(R4S)系统作为国际协作的新生儿筛查数据平台(
2013年10月至2018年7月,浙江省新生儿疾病筛查中心应用串联质谱技术筛查新生儿2 040 072名,经切值法判读910例为疑似VLCADD阳性。本研究通过浙江大学医学院附属儿童医院伦理委员会批准(批准号:2018-IRB-077)。
1. 血片采集和串联质谱技术(MS/MS)测定:按照《新生儿疾病筛查技术规范》(2010年版),新生儿出生满72 h且充分哺乳6次后,由专业医护人员采集足跟血滴于干净Schleicher & Schuell 903滤纸上,形成3个直径≥8 mm的血斑,室温避光自然干燥[7]。滤纸血片通过冷链物流系统递送至浙江省新生儿疾病筛查中心实验室,应用串联质谱法进行集中检测,测定仪器为AQCUTY TQD/XEVO-TQD串联质谱仪(Waters公司,美国),试剂为NeoBase非衍生试剂盒(PerkinElmer公司,芬兰)。
2. 切值法判读:切值由频数分布法计算600 000名健康新生儿99.5%的百分位数设定。十四烯酰基肉碱(C14∶1)高于切值或相关比值升高者,进行原血片复查,2次检测结果经临床医生综合分析判断,将初筛疑似VLCADD阳性患儿召回复查MS/MS,进一步临床检测,必要时进行基因确诊;当C14∶1显著升高,疑似患儿应立即召回确诊。
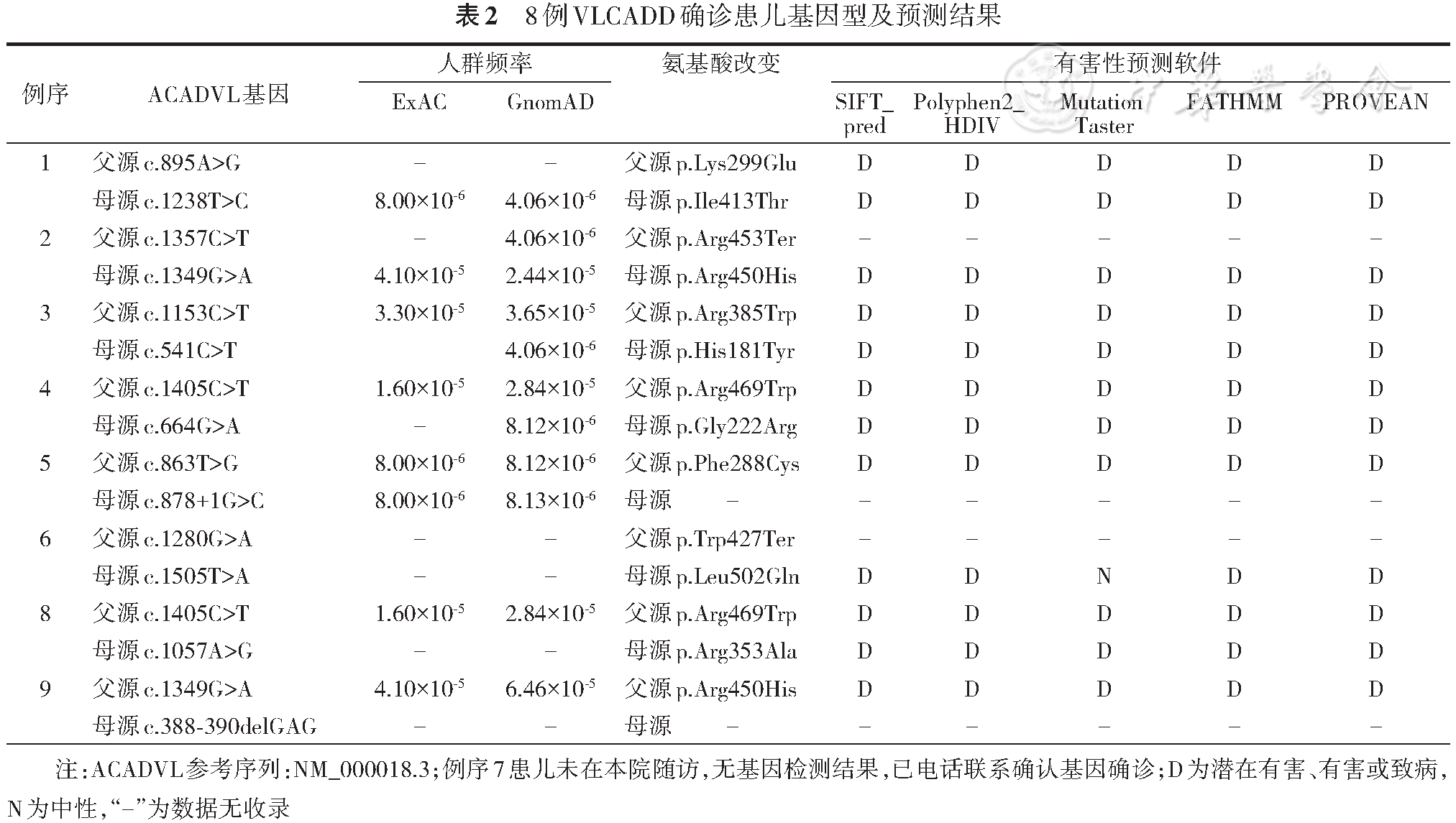
3. 基因检测:采集疑似VLCADD阳性患儿及其父母的外周血各2 m1(乙二胺四乙酸二钠抗凝),使用二代高通量靶向测序进行ACADVL基因检测,测序结果与1000基因组数据库、ESP6500数据库、ExAC数据库、GnomAD数据库进行比对,获得可疑的突变位点。应用SIFT_pred、Polyphen2_ HDIV、Mutation Taster、FATHMM、PROVEAN在线软件进行变异位点对蛋白质功能影响有害性预测。根据美国医学遗传学与基因组学学会(American College of Medical Genetics and Genomics,ACMG)序列变异解释指南[8]对变异位点进行致病性分析。对明确基因变异患儿的父母进行Sanger测序,确定变异来源。
4. 联合R4S系统分析:(1)R4S系统数据处理:将经串联质谱筛查切值法判读疑似VLCADD阳性患儿原始数据转换为CSV格式文件上传至R4S系统平台,应用VLCADD分析工具进行分析。(2)VLCADD分析工具:为R4S系统单疾病分析工具之一,由VLCADD特异性指标群组成的累积评分系统,包括C14∶1、十四二烯酰基肉碱(C14∶2)、十四烷酰基肉碱(C14)、十二烯酰基肉碱(C12∶1)、十六烷酰基肉碱(C16)、乙酰基肉碱(C2)的浓度和C14∶1/C16、C14∶1/C2、C14∶1/C12∶1等。VLCADD分析工具对样本的上述指标数值与数据库正常范围及疾病范围的重合程度计算得分,依据得分给出相应结论以标识不同程度VLCADD阳性可能,分别为无意义、可能、很可能及极可能,除无意义外,均提示阳性。
5. C14∶1人群分布百分位数比较:计算浙江省新生儿疾病筛查中心C14∶1浓度值的5个百分位数(1%、10%、50%、90%和99%),上传至R4S系统平台,与R4S数据库相应百分位数比较分析,并根据操作指南,由R4S系统判断VLCADD单疾病工具是否适用于浙江省新生儿人群分布。
6. 统计学分析:使用SPSS 22.0统计软件进行统计学分析。以临床诊断及ACADL基因检测结果为标准,依据R4S操作指南分别计算单纯应用切值法与联合R4S系统的假阳性率(筛查假阳性/筛查总量)、阳性预测值(真阳性/筛查阳性)、敏感度[真阳性/(真阳性+假阴性)]、特异度[真阴性/(真阴性+假阳性)]。组间率的比较采用χ?检验,P<0.05为差异有统计学意义。
浙江省新生儿疾病筛查中心自2013年10月至2018年7月采用串联质谱(非衍生法)筛查新生儿共2 040 072名,切值法判读疑似VLCADD阳性患儿910例(召回903例,失访7例),召回率99.23%。其中基因检测确诊9例,患病率为1/226 674,假阳性率0.44‰,阳性预测值0.99%,特异度99.96%,敏感度100%,除1例为早产儿(孕36周),其余均为足月正常体重儿。
第9例确诊患儿相关串联指标检测结果明显异常(
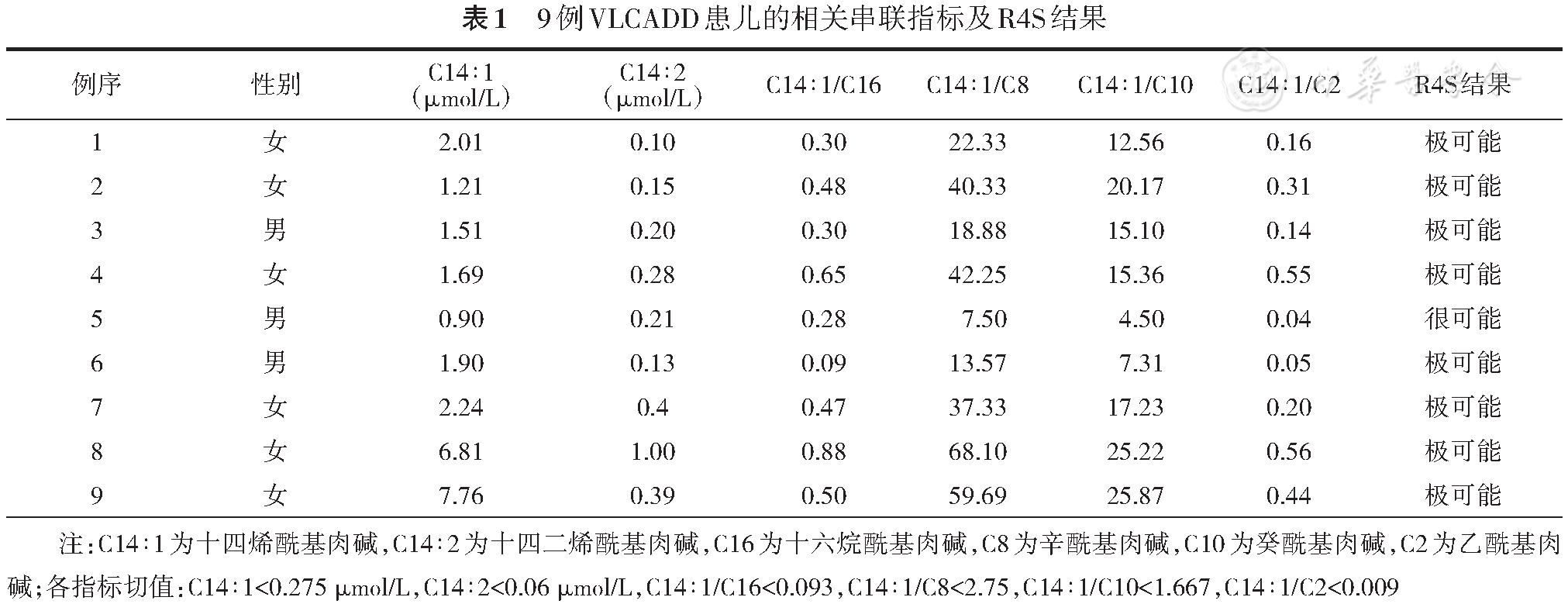
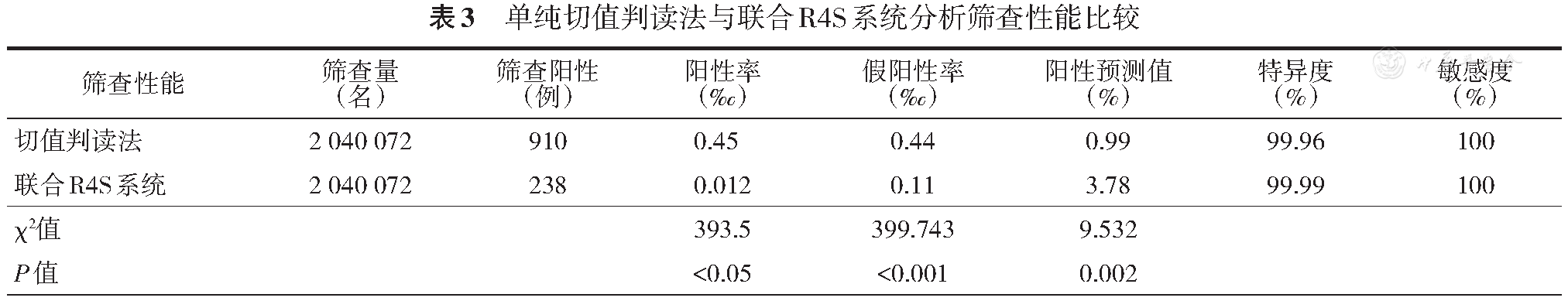
R4S系统VLCADD单疾病工具对910例VLCADD初筛疑似阳性样本的串联质谱数据分析显示,238例提示阳性(含基因确诊9例),排除假阳性672例。比较单纯切值法与联合R4S系统分析的各筛查性能指标,提示2种方法的阳性率、假阳性率及阳性预测值差异有统计学意义(P<0.05),敏感度和特异度是基于真假阳性计算的比值,不单列统计学分析(
浙江省人群C14∶1的5个百分位数(1%、10%、50%、90%、99%)分别为0.03、0.05、0.10、0.20和0.32 μmol/L,R4S系统数据库C14∶1的5个百分位数(1%、10%、50%、90%、99%)分别为0.03、0.05、0.08、0.15和0.26 μmol/L;二者1%和10%相同,50%相近,90%和99%差异<25%。根据R4S操作指南定义,对于浙江省新筛C14∶1人群分布情况,R4S系统VLCADD单疾病工具可适用性良好。
VLCADD发病率较低,在不同国家和地区存在明显差异[9]。欧美国家VLCADD发病率在1/10万~1/3万,为第二大最常见的脂肪酸代谢性疾病[10, 11]。Shibata等[12]分析亚洲各国使用MS/MS进行新生儿VLCADD筛查的数据,日本1997年至2015年336万新生儿的发病率在1/93 000,韩国2000年至2015年344万新生儿的发病率在1/38 300,我国台湾地区发病率约为1/1 392 000,其他地区发病率尚不明确。浙江省新生儿疾病筛查中心检测2 040 072例新生儿,确诊VLCADD患儿9例,发病率约为1/226 674。
根据发病年龄和临床症状,VLCADD可分为3个类型:心肌病型(新生儿和婴儿早期)、肝型(婴儿后期及儿童)和肌病型(青少年或成年)。其中心肌病型发病最凶险,患儿死亡率高[13],通过新生儿筛查及早诊断并干预治疗能有效提高该型VLCADD患儿的生存率,也能改善其他两型患儿的预后。目前发达国家和我国新生儿筛查实验室多采用串联质谱技术(MS/MS)检测以C14∶1为主的相关指标进行VLCADD筛查[14, 15],该法虽具有高特异度和敏感度的优点,但基于目前判读缺乏标准化和规范化的现状,不同人员依据个人经验对同一检测结果的解读存在差异等因素,存在假阳性率较高,阳性预测值偏低的缺点[16]。浙江省新生儿疾病筛查中心单纯使用切值法判读MS/MS结果,筛查近40种遗传代谢病,VLCADD的初筛阳性率位列前五,假阳性率位居前三。对于以检测丙酰基肉碱(C3)、异戊酰基肉碱(C5)等为特异性指标的遗传代谢病,可采用二阶筛查方法增加阳性预测值[17],但C14∶1指标目前缺乏二阶筛查方法;虽然根据孕周体重单独设定切值能有效降低假阳性率[18],但操作复杂,临床实用性不高。因此,提高筛查性能与疾病诊断的准确性、降低假阳性率,是当前开展新生儿VLCADD筛查亟待解决的问题。
R4S项目通过批量处理新筛数据、验证可疑阳性病例、评估现用切值、优化临床特异性指标和自动提示疾病类型等方式,改善新筛中假阳性偏高等问题,提高筛查效率。运用R4S系统实例显示,瑞士18万新生儿筛查假阳性率由0.07%降至0.05%、意大利近10万新生儿筛查假阳性率下降1.7%、美国多州新生儿筛查假阳性率下降显著[19]。Fleischman等[20]认为R4S部分单疾病工具在一定程度上可增加检出阳性病例,减少假阳性率。由此可见,R4S系统能有效降低新生儿筛查假阳性率,增强新生儿筛查效率。
然而,国内胡真真等[6]应用R4S系统对362 822例新生儿串联质谱数据进行分析,结果显示因人群分布差异导致大量阴性样本仍需结合传统判断规则进行排除,建议应用时应结合本地健康人群范围进一步优化;同时β酮硫解酶缺乏症及脯氨酸血症等有误诊和漏诊现象。Merritt等[21]验证52例VLCADD确诊病例,其中R4S提示阳性的只有15例,其漏诊原因可能与当时数据库阳性病例积累不足有关。目前R4S系统已收录VLCADD确诊阳性192例,且提示浙江省C14∶1人群分布与数据库相近,本次研究应用切值法联合R4S系统判读,假阳性率显著降低且无漏诊。
综上所述,R4S系统联合切值法应用于串联质谱VLCADD新生儿筛查可显著提高筛查性能,降低假阳性率,具有一定的临床应用价值,是解决我国新筛现存问题的新方法。国外虽已有多篇文献报道应用R4S系统可提升新生儿筛查项目的工作效率,但国内对R4S系统的应用研究较少。由于人群差异和某些疾病数据库阳性病例积累不足等局限性因素,对于其他新生儿遗传代谢类疾病的应用,还需联合切值法以及临床综合判断等进一步研究和优化。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
