非小细胞肺癌靶向治疗主要靶点及其检测方法进展
2020年8月
中华神经科杂志,第43卷第8期 第842页-第848页
邵璇璇,高春芳,贾健安
在全球范围内,肺癌的发病率与死亡率已成为恶性肿瘤首位[1],2015年中国癌症统计显示,我国肺癌发病与死亡人数分别为73.33万和61.02万例[2]。根据免疫组化将肺癌分为小细胞肺癌和非小细胞肺癌(non-small cell lung cancer, NSCLC),其中NSCLC约占肺癌80%~85%[3]。现已证实,驱动基因突变是NSCLC的最主要致病机制之一[4],其中最主要的突变基因包括受体酪氨酸激酶(receptor tyrosine kinases, RTK)家族及其下游信号通路,特别是RAS-RAF-MAPK通路及PI3K-AKT通路[5]。临床上广泛使用的晚期NSCLC靶向治疗药物也正是靶向上述驱动基因突变,因此相关基因突变的检测也成为指导靶向治疗的重要原则,这种为明确特定治疗手段安全性与有效性的体外诊断方法即为伴随诊断[6],目前,用于指导NSCLC靶向治疗最常检测的基因包括:表皮生长因子受体(epidermal growth factor receptor, EGFR)、间变性淋巴瘤激酶(anaplastic lymphoma kinase,ALK)、c-ros原癌基因1酪氨酸激酶(proto-oncogene tyrosine-protein kinase,ROS1)、鼠类肉瘤病毒癌基因(KRAS)、B-Raf原癌基因丝/苏氨酸激酶(B-Raf proto-oncogene, serine/threonine kinase,BRAF)等[7,8,9],此外,人表皮生长因子受体-2(human epidermal factor receptor,Her2)、磷脂酰肌醇-3激酶(phosphatidylinositol-3 kinases,PI3K)、RET原癌基因(rearranged during transfection proto-oncogene,RET)和间质上皮转化基因(the mesenchymal-to-epithelial transition,MET)等基因的检测也逐渐进入临床应用[10]。
上述基因通过多种变异影响肿瘤进展及靶向治疗效果,变异形式主要包括:点突变、插入/缺失、重排/融合、扩增等,常用的检测方法包括实时荧光定量PCR法、免疫组化、荧光原位杂交、基因测序等,同时随着用于临床的靶向治疗药物越来越多,如何针对特定基因变异类型选择合适的检测方法,也成为NSCLC靶向治疗伴随诊断领域需要关注的重要问题之一。
本研究根据中国国家药品管理监督局(National Medical Products Administration, NMPA)中NSCLC的常见靶点基因突变及其检测方法进行归纳整理(试剂盒有效数据均统计到2019年12月1日),为临床选择适合于NSCLC伴随诊断(基因突变检测方法与试剂)提供一定依据,也为临床医师基于合理有效的伴随诊断方案实施个体化治疗提供支持。
EGFR基因是人表皮生长因子受体家族成员之一,是一种跨膜酪氨酸激酶型受体,可被细胞外配体激活并向下游传递信号,促进细胞增殖等功能[11]。在EGFR突变中,18~21号外显子突变率占EGFR常见突变中的95%以上[11],其中第19号外显子非移码缺失突变(Del19)和21号外显子的点突变(L858R)为敏感突变,约占84.60%,其他非经典突变约占15.4%,带有EGFR Del19及L858R此类敏感突变的NSCLC患者对酪氨酸激酶抑制剂类药物(tyrosine kinase inhibitors, TKI)治疗敏感[12]。
目前,临床上多采用荧光原位杂交技术(fluorescence in situ hybridization, FISH)检测EGFR的基因扩增,荧光PCR法和测序法检测基因突变。测序法有Sanger测序和下一代测序(next-generation sequencing,NGS)。市面上EGFR的检测试剂与方法多种多样,NMPA所列的EGFR基因突变检测试剂汇总如

将检测EGFR基因突变的荧光PCR方法根据标本类型及检测突变基因种类进行分析,如

现已证实,NSCLC患者对TKIs药物的敏感性与EGFR基因突变有关[13],研究表明EGFR扩增与TKIs药物治疗后患者的无病生存期和总生存期密切相关[14],同时检测EGFR基因扩增和突变可以更准确反映EGFR基因状态并指导TKIs用药。EGFR基因突变类型与种类众多,报道显示EGFR基因突变类型多达39种[11],并且仍有新的突变类型不时见于报道[15,16],可见EGFR基因的突变位点与突变类型的多样性,对于复杂的19外显子突变位点与突变类型,实时荧光PCR方法容易造成漏检,此时选用测序法比较适宜。而18、20外显子突变频率相对19与21外显子较低,所以在临床应用时应该增加对18、20外显子突变的检测以提高EGFR基因突变检测能力,指导NSCLC患者的靶向治疗有效性。另外,EGFR基因突变存在着显著的地域、种族差异[17],在不同地区采用同一检测方法可能会出现不同的突变检出率,因此在考虑选择EGFR基因突变检测方法时,方法学性能与基因突变分布特点均应考虑在内,并应根据种族构成选择检测方法。
带有EGFR突变的NSCLC患者对初始TKI药物治疗应答良好,但一部分患者在平均治疗10个月左右会出现耐药现象[18],其中以EGFR T790M突变最为常见[19],对于一代TKI耐药患者,第三代TKI可以显著延长T790M突变的晚期NSCLC患者的无进展生存期[20],虽然组织标本EGFR T790M检测是金标准,但对于晚期NSCLC患者,组织标本获取不易且数量有限,部分患者由于组织标本不足或无法获取组织标本而无法进行分子检测,TKI治疗进展的NSCLC患者可进行组织再次活检的比例不足50%,而血液标本无创且可重复采样,使用血液标本进行EGFR基因检测可以帮助患者有效节省有限的组织标本,扩大受益人群,实现精准诊疗的较大获益。因此,对于一代TKI治疗失败后发生T790M突变的患者,可以首先选择液体活检T790M突变检测来评估是否可以接受第三代TKI药物治疗,如外周血检测阴性,可以考虑进一步做组织活检检测。目前,NMPA批准的大部分EGFR变异荧光PCR试剂均可检测石蜡包埋组织的EGFR T790M突变(
ALK基因位于2号染色体,编码一种跨膜受体酪氨酸激酶,其变异形式包括过表达、点突变、与其他基因融合表达等,ALK基因融合突变是NSCLC常见的一种驱动基因,能恒定激活下游信号通路,导致致癌驱动基因的异常表达[21]。临床上多用ALK激酶抑制剂如克唑替尼治疗ALK基因融合患者,能够显著延长生存期[22]。
目前NMPA批准了11种试剂可用于NSCLC患者的ALK基因融合检测,检测方法包括:测序、荧光原位杂交法、免疫组化和荧光PCR,如
ROS1是一种原癌基因,位于6号染色体上,与EGFR、ALK同属受体酪氨酸激酶家族,编码胰岛素受体的一种,NSCLC患者中ROS1常与其他基因发生融合,从而持续激活下游信号,促进细胞生长与增殖[23]。ROS1常在年轻、不抽烟或轻度抽烟的患者中检出,一系列的临床研究表明克唑替尼对ROS1阳性的进展期肺癌患者治疗高度有效[24]。NMPA目前仅批准了4种ROS1基因重排检测试剂用于NSCLC患者伴随诊断,包括荧光PCR方法和测序法(
KRAS基因位于12号染色体,编码一种细胞膜内侧的膜结合型蛋白,位于受体酪氨酸激酶信号通路的下游,介导RAS-RAF-MAPK信号通路,对肿瘤的发生发展至关重要,KRAS最常见的突变位于外显子1的第12和13位氨基酸,以及外显子2的第61位氨基酸,KRAS基因发生突变引起多种增殖因子被激活,直接激活下游信号,不依赖上游信号,因此导致多种TKI治疗无效[25],故对KRAS基因突变进行检测可用于指导TKI应用,并辅助NSCLC患者预后判断[26]。
NMPA批准的KRAS基因突变检测试剂有13种,批准试剂数量仅次于EGFR变异检测试剂数量,可见其在NSCLC伴随诊断中的重要性,采用的方法包括荧光PCR方法、基因芯片法、流式荧光杂交法和测序法(
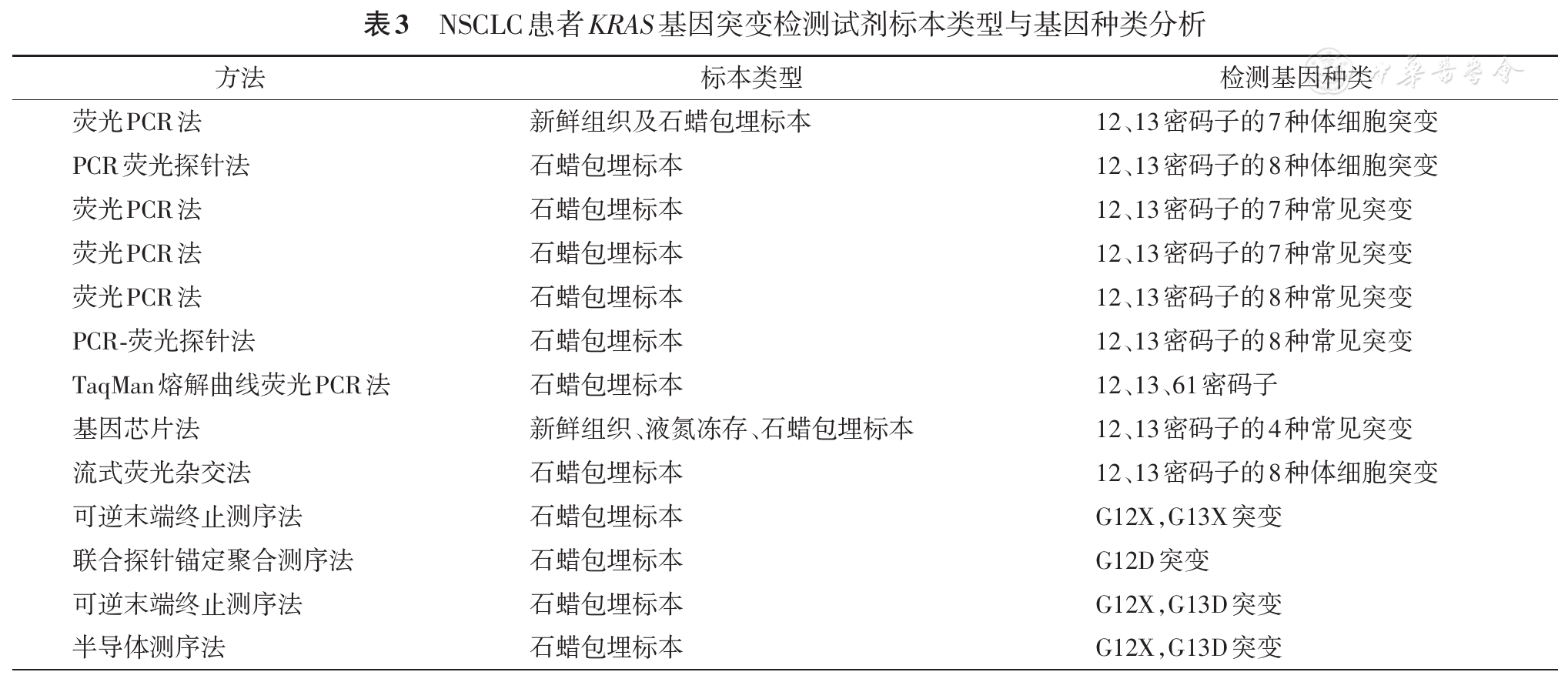
BRAF基因位于7号染色体,编码一种苏氨酸蛋白激酶,参与RAS-RAF-MAPK信号通路,是RAS的下游信号分子。BRAF最常见的突变位点位于第600位氨基酸,即V600E突变,也是目前临床最主要检测的突变位点,携带BRAF V600E的NSCLC患者对威罗菲尼治疗敏感[27]。NMPA批准了11种BRAF基因突变检测试剂用于NSCLC患者伴随诊断(
临床上除了检测上述基因外,还有其他一些基因也用于指导NSCLC靶向治疗,主要包括:Her2、PI3KCA、RET、MET等。
Her2与EGFR同属人表皮生长因子受体家族成员之一,细胞膜表面Her2过表达会导致下游信号通路持续激活,促进肿瘤增殖[28],NSCLC患者Her2过表达或基因拷贝数高,与TKIs获得性耐药有关[29],根据NCCN非小细胞肺癌临床实践指南,Her2突变患者可考虑使用抗体耦联曲妥珠单抗进行治疗。Her2过表达可通过免疫组化方法进行检测,FISH可用于检测肿瘤细胞中Her2拷贝数,目前NMPA批准了9种FISH试剂、两种免疫组化试剂及两种NGS试剂可用于检测Her2表达水平或拷贝数变化,但主要用于乳腺癌Her2基因检测。
PI3KCA是PI3K的一种,其编码的激酶接受来自细胞膜RTK的信号,介导PI3K-AKT信号通路,PI3KCA基因突变或过表达会导致该通路持续激活[34],从而促进增殖,相比腺癌患者,肺鳞癌患者中PI3KCA基因突变或过表达比率更高,可达10%~30%[35],PI3KCA基因突变常与TKIs获得性耐药有关,且与患者预后不良有关[36]。目前NMPA已批准了FISH、荧光PCR及NGS试剂可用于PI3KCA突变或过表达的检测。
RET原癌基因位于10号染色体,编码一种RTK,可激活下游信号通路,肿瘤细胞中RET可与KIF5B等基因发生融合表达[32],持续激活下游信号,促进细胞增殖。带有RET重排的NSCLC患者可选择卡博替尼进行治疗[33]。目前NMPA仅批准了NGS方法用于RET基因重排的检测。
MET也编码一种RTK,正常情况下,可与泛素化蛋白结合被降解,发生第14号外显子缺失的met蛋白可不被降解,从而持续激活,促进肿瘤生长[30],携带MET 14号外显子缺失的患者,可使用克唑替尼进行治疗[31],目前NMPA仅批准了NGS方法用于MET基因跳跃(MET exon 14 skipping)的检测。
随着NGS技术的进步,在临床诊疗过程中基因检测在NSCLC患者治疗中日益广泛,从最初的单基因检测发展到多基因检测,使得NSCLC更加趋向细化诊断、精准治疗,也为NSCLC的诊疗提供了更多的可能性。目前NMPA批准了四种测序试剂盒可用于NSCLC的伴随诊断,现将四种试剂盒的检测基因、标本类型、测序平台、检测限、靶向药物等归纳分析见
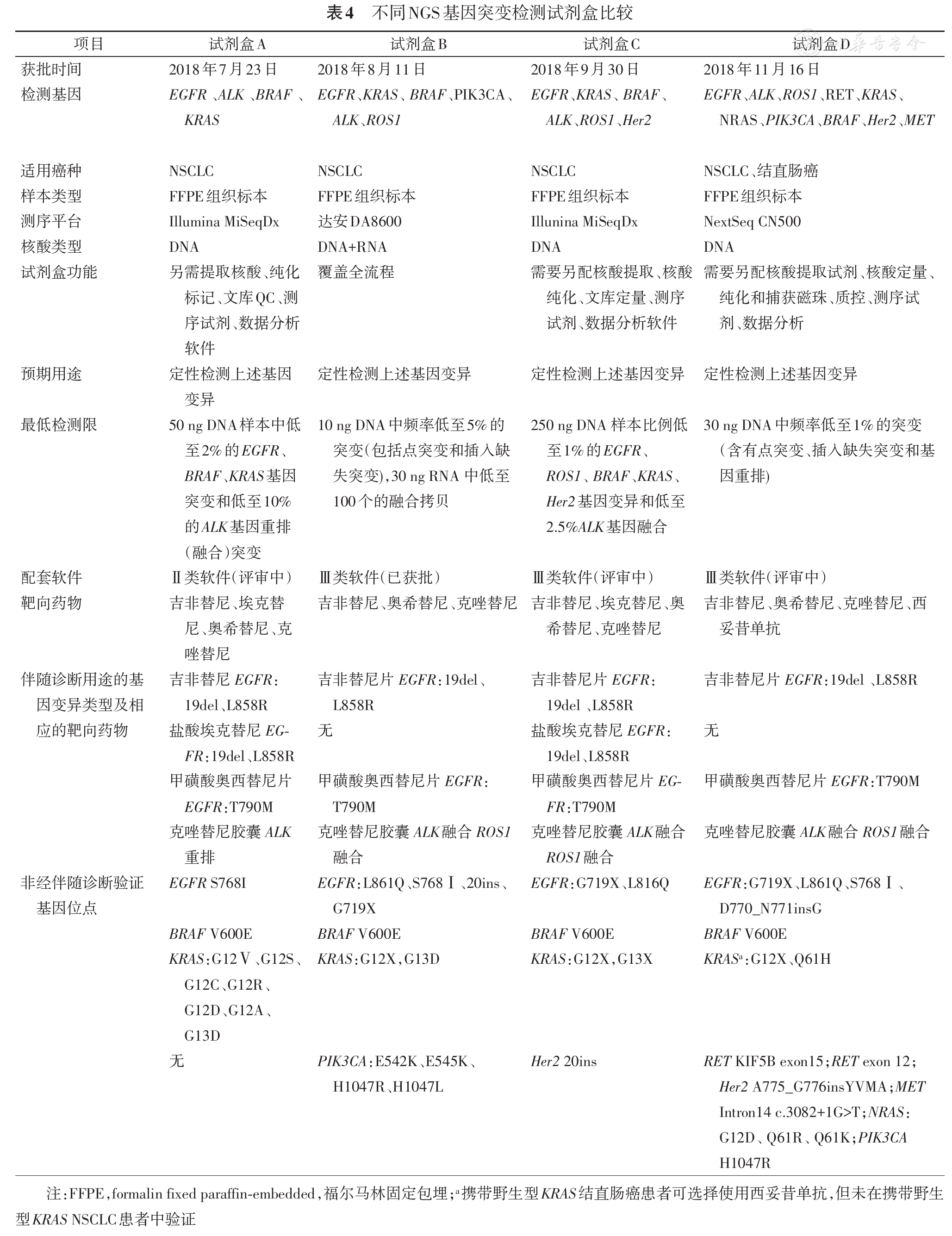
目前市面上的四种用于NSCLC的NGS基因检测试剂盒主要检测EGFR、ALK、BRAF、KRAS、ROS1等基因,适用于不同的基因测序仪,因此,不同试剂盒检测结果之间难以直接比较。上述试剂盒均提供了较多的未经伴随诊断验证基因位点的变异信息,这也是NGS技术的优势。上述部分试剂盒还需要另外配套核酸提取、质控等试剂。由于NGS的数据量大,数据分析过程较复杂,目前仅有一种试剂盒配套分析软件已经获批,其余三种试剂盒的配套分析软件仍在审批中,这也为上述NGS试剂盒的临床规范应用带来了一定困难。
目前NMPA批准的适用于NSCLC相关基因变异检测的方法较多,各有优缺点,实时荧光PCR方法步骤简单快捷,实验耗时比较短,因此获NMPA批准试剂数量最多,但只能检测已知突变位点与类型,适合少数突变位点或类型的检测。免疫组化方法因其具有简便易行、操作方法成熟等优点应用广泛,但其灵敏度低,不能明确融合基因型。FISH能比较特异地检出基因融合及基因重排,但是操作繁琐、价格较高,检测结果主观性较强,规范判读要求较高。免疫组化与FISH对于基因大片段变化检测较为适用,对于点突变等微小改变检测敏感性较低。Sanger测序法目前是基因突变检测的金标准方法,可检测已确定的和一些未知的突变位点,结果比较可靠,重复性好,但通量有限,灵敏度有限。NGS检测通量高,灵敏度高,可检测未知突变位点,适合多个基因组合检测,更适合指导NSCLC患者的靶向药物治疗,但同时对样本的要求也比较高,实验步骤多,耗时长,同时市面上可以选择的检测试剂盒较为有限,数据分析要求较高。各实验室应该根据自身情况,选择一种检测平台作为基础,其他方法用于验证,可以更准确的反映基因状态,更好的指导个体化治疗方案的选择及患者预后评估。
随着分子靶向治疗的不断进展,越来越多的靶向药物投入临床应用,相应伴随诊断的成本不断下降,多基因联合检测将成为未来NSCLC伴随诊断的主流筛选手段,可以更加有效的筛选目标人群,更好的选择治疗时机,不断提高靶向治疗效果。
[1] 郑荣寿,孙可欣,张思维,等. 2015年中国恶性肿瘤流行情况分析[J].中华肿瘤杂志, 2019, 41(1): 19-28.
[2] Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. Ca Cancer J Clin, 2016, 66(2): 115-132.
[3] Pikor LA, Ramnarine VR, Lam S, et al. Genetic alterations defining NSCLC subtypes and their therapeutic implications[J]. Lung Cancer, 2013, 82(2): 179-189.
[4] Zehir A, Benayed R, Shah RH, et al. Mutational Landscape of Metastatic Cancer Revealed from Prospective Clinical Sequencing of 10,000 Patients[J]. Nat Med, 2017, 23(6): 703-713.
[5] Heist RS, Engelman JA. SnapShot: non-small cell lung cancer[J]. Cancer cell, 2012, 21(3): 448.
[6] U. S. Food And Drug Administration. Guidance for Industry: In Vitro Companion Diagnostic Devices[J]. 2014.
[7] Solomon B, Mok T, Kim D, et al. First-line crizotinib versus chemotherapy in
[8] Shaw AT, Ou SI, Bang Y, et al. Crizotinib in
[9] Lievre A, Bachet J, Corre DL, et al.
[10] Guo Y, Cao R, Zhang X, et al. Recent Progress in Rare Oncogenic Drivers and Targeted Therapy For Non-Small Cell Lung Cancer[J]. Onco Targets Ther, 2019, 12: 10343-10360.
[11] Sharma SV, Bell DW, Settleman J, et al. Epidermal growth factor receptor mutations in lung cancer[J]. Nat Rev Cancer, 2007, 7(3): 169-181.
[12] Russo A, Franchina T, Ricciardi GRR, et al. Heterogeneous Responses to Epidermal Growth Factor Receptor (EGFR) Tyrosine Kinase Inhibitors (TKIs) in Patients with Uncommon EGFR Mutations: New Insights and Future Perspectives in this Complex Clinical Scenario[J]. Int J Mol Sci, 2019, 20(6): 1431.
[13] Lee CK, Brown C, Gralla RJ, et al. Impact of EGFR Inhibitor in Non-Small Cell Lung Cancer on Progression-Free and Overall Survival: A Meta-Analysis[J]. J Natl Cancer Inst, 2013, 105(9): 595-605.
[14] Yatabe Y, Takahashi T, Mitsudomi T. Epidermal Growth Factor Receptor Gene Amplification Is Acquired in Association with Tumor Progression of EGFR-Mutated Lung Cancer[J]. Cancer Res, 2008, 68(7): 2106-2111.
[15] Maignan LL, Mirebeau-Prunier D, Vervueren L, et al. First Case of A859T Epidermal Growth Factor Receptor Mutation Responding to Erlotinib[J]. J Thorac Oncol, 2011, 6(3): 639-640.
[16] Costa DB, Schumer ST, Tenen DG, et al. Differential Responses to Erlotinib in Epidermal Growth Factor Receptor (EGFR)-Mutated Lung Cancers With Acquired Resistance to Gefitinib Carrying the L747S or T790M Secondary Mutations[J]. J Clin Oncol, 2008, 26(7): 1182-1186.
[17] Huang SF, Liu H, Li L, et al. High frequency of epidermal growth factor receptor mutations with complex patterns in non-small cell lung cancers related to gefitinib responsiveness in Taiwan.[J]. Clin Cancer Res, 2004, 10(24): 8195-8203.
[18] Ercan D, Choi HG, Yun CH, et al. EGFR Mutations and Resistance to Irreversible Pyrimidine-Based EGFR Inhibitors[J]. Clin Cancer Res, 2015, 21(17).
[19] Camidge DR, Pao W, Sequist LV. Acquired resistance to TKIs in solid tumours: learning from lung cancer[J]. Nat Rev Clin Oncol, 2014, 11(8): 473-481.
[20] Mok T, Wu Y, Ahn M, et al. Osimertinib or Platinum-Pemetrexed in EGFR T790M-Positive Lung Cancer[J]. N Engl J Med, 2017, 376(7): 629-640.
[21] Qian M, Zhu B, Wang X, et al. Drug resistance in ALK-positiveNon-small cell lungcancer patients[J]. Semin Cell Dev Biol, 2016, 64: S427660587.
[22] Solomon BJ, Mok T, Kim D, et al. First-line crizotinib versus chemotherapy in ALK-positive lung cancer[J]. N Engl J Med, 2014, 371(23): 2167-2177.
[23] Giulio R, Genny J, Antonia C, et al. Detection of ROS1 rearrangement in non-small cell lung cancer: current and future perspectives[J]. Lung Cancer (Auckl), 2017, Volume 8∶45-55.
[24] Wu Y, Yang JC, Kim D, et al. Phase II Study of Crizotinib in East Asian Patients With ROS1-Positive Advanced Non-Small-Cell Lung Cancer[J]. J Clin Oncol, 2018, 36(14): 1405-1411.
[25] Murugan AK, Grieco M, Tsuchida N. RAS mutations in human cancers: Roles in precision medicine[J]. Semin Cancer Biol, 2019,59:23-35.
[26] 刘蕾,魏素菊. KRAS突变的非小细胞肺癌的研究进展[J].中国肺癌杂志, 2018, 21(5): 419-424.
[27] Zaman A, Wu W, Bivona TG. Targeting Oncogenic BRAF: Past, Present, and Future[J]. Cancers, 2019, 11(8): 1197.
[28] Hervent AS, De Keulenaer GW. Molecular Mechanisms of Cardiotoxicity Induced by ErbB Receptor Inhibitor Cancer Therapeutics[J]. Int J Mol Sci, 2012, 13(12): 12268-12286.
[29] Takezawa K, Pirazzoli V, Arcila ME, et al. HER2 Amplification: A Potential Mechanism of Acquired Resistance to EGFR Inhibition in EGFR-Mutant Lung Cancers That Lack the Second-Site EGFRT790M Mutation[J]. Cancer Discovery, 2012, 2(10): 922-933.
[30] Cipriani NA, Abidoye O, Vokes EE, et al. MET as a target for treatment of chest tumors[J]. Lung Cancer, 2009, 63(2): 169-179.
[31] Cortot AB, Kherrouche Z, Descarpentries C, et al. Exon 14 Deleted MET Receptor as a New Biomarker and Target in Cancers.[J]. J Natl Cancer Inst, 2017, 109(5).
[32] Wang R, Hu H, Pan Y, et al. RET Fusions Define a Unique Molecular and Clinicopathologic Subtype of Non-Small-Cell Lung Cancer[J]. J Clin Oncol, 2012, 30(35): 4352-4359.
[33] Mendoza L. Clinical development of RET inhibitors in RET-rearranged non-small cell lung cancer: Update[J]. Oncol Rev, 2018, 12(2):352.
[34] Dienstmann R, Rodon J, Serra V, et al. Picking the Point of Inhibition: A Comparative Review of PI3K/AKT/mTOR Pathway Inhibitors[J]. Mol Cancer Ther, 2014, 13(5): 1021-1031.
[35] Zheng D, Wang R, Ye T, et al. MET exon 14 skipping defines a unique molecular class of non-small cell lung cancer[J]. Oncotarget, 2016, 7(27): 41691-41702.
[36] Sequist LV, Waltman BA, Diassantagata D, et al. Genotypic and Histological Evolution of Lung Cancers Acquiring Resistance to EGFR Inhibitors[J]. Sci Transl Med, 2011, 3(75).
